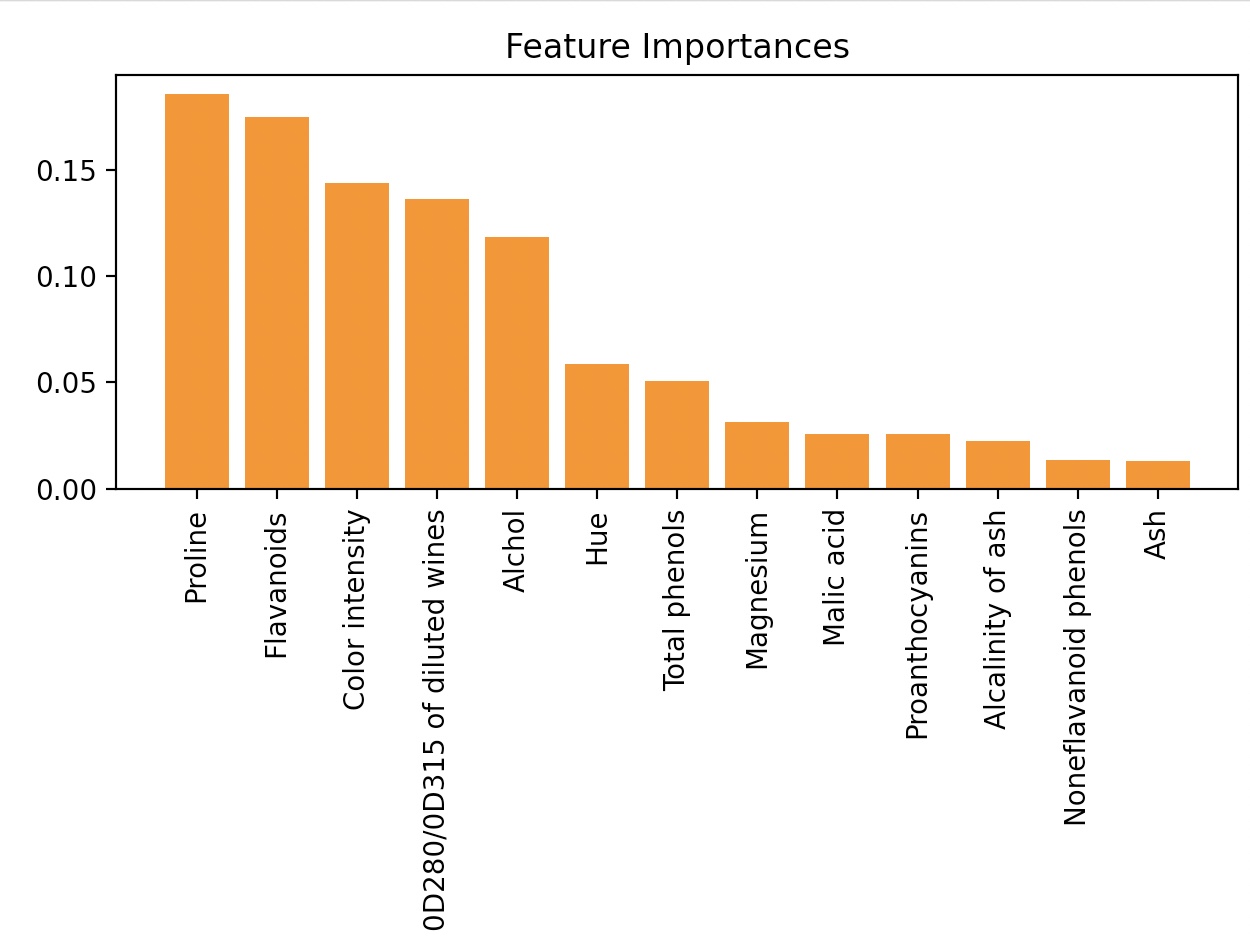
Wine 데이터셋에서 500개의 트리를 가진 랜덤 포레스트를 구현하고 중요도에 따라 13개의 특성에 순위를 매긴다.
from sklearn.ensemble import RandomForestClassifier
feat_labels=df_wine.columns[1:]
forest=RandomForestClassifier(n_estimators=500, random_state=1)
forest.fit(X_train, y_train)
importances=forest.feature_importances_
indices=np.argsort(importances)[::-1]
for f in range(X_train.shape[1]):
print("%2d)%-*s %f" %(f+1, 30, feat_labels[indices[f]], importances[indices[f]]))
plt.title('Feature Importances')
plt.bar(range(X_train.shape[1]), importances[indices], align='center')
plt.xticks(range(X_train.shape[1]), feat_labels[indices], rotation=90)
plt.xlim([-1, X_train.shape[1]])
plt.tight_layout()
plt.show()
1)Proline 0.185453
2)Flavanoids 0.174751
3)Color intensity 0.143920
4)0D280/0D315 of diluted wines 0.136162
5)Alchol 0.118529
6)Hue 0.058739
7)Total phenols 0.050872
8)Magnesium 0.031357
9)Malic acid 0.025648
10)Proanthocyanins 0.025570
11)Alcalinity of ash 0.022366
12)Noneflavanoid phenols 0.013354
13)Ash 0.013279
Wine 데이터셋에서 특성의 상대적인 중요도에 따른 순위를 그래프로 나타낸다.
특성 중요도 합은 1이 되도록 정규화 된 값이다.
랜덤 포레스트에서 두 개 이상의 특성이 매우 상관관계가 높다면, 하나의 특성은 매우 높은 순위를 갖지만,다른 특성 정보는 완전히 잡아내지 못할 수도 있다.
이는 특성 중요도 값은 해석하는 것보다 모델의 예측 성능에만 관심을 가진다면, 신경 쓸 필요는 없다.
SelectFromModel
SelectFromModel은 Pipeline의 중간 단계에서 RandomForestClassifier를 특성 선택기로 사용할 때 유용하다.
( 모델 훈련이 끝난 후에 사용자가 지정한 임계 값을 기반으로 특성을 선택한다.
coef_ , feature_importances_ 속성과 임계 값을 비교해서 특성을 선택한다.
importance_getter 매개변수가 사이킷런 024버전에서 추가되었다. (비교할 속성을 지정)
threshold 매개변수로 임계값을 지정할 수 있다. median mean으로 지정하여 중간값이나 평균값 사용
기본값은 mean이다.(아래 예제에서는 0.1 이상)
from sklearn.feature_selection import SelectFromModel
sfm=SelectFromModel(forest, threshold=0.1, prefit=True)
X_selected=sfm.transform(X_train)
print('이 임계 조건을 만족하는 샘플의 수:', X_selected.shape[1])
원래는 fit() 메서드를 이용해서 훈련시킨다. 앞에서 랜덤 포레스트 모델을 이미 훈련하였기에 위에서는 tranform() 메서드를 바로 사용
for f in range(X_selected.shape[1]):
print("%2d) %-*s %f" %(f+1, 30, feat_labels[indices[f]], importances[indices[f]]))
1) Proline 0.185453
2) Flavanoids 0.174751
3) Color intensity 0.143920
4) 0D280/0D315 of diluted wines 0.136162
5) Alchol 0.118529
재귀적 특성 제거(RFE)
처음에 모든 특성을 사용하여 모델을 만들고, 특성 중요도가 낮은 특성을 제거한다.
그 다음 제외된 특성을 빼고 새로운 모델을 만들고 미리 정의한 특성 개수가 남을 때까지 이를 반복한다.
n_feature_to_select 매개변수를 통해 선택할 특성의 개수를 지정 (0, 1)사이의 숫자를 통해 비율 사용 가능
step 매개변수를 통해 각 반복에서 제거할 특성의 개수를 지정한다. (0, 1)사이의 숫자를 통해 삭제할 특성의 비율 사용 가능
defalut값은 1이다.
기본적으로 coef_ , feature_importances_ 속성을 기준으로 특성을 제거한다.
사이킷런 0.24 버전에서 사용할 속성을 지정할 수 있는 importance_getter 매개변수가 추가되었다.
from sklearn.feature_selection import RFE
rfe=RFE(forest, n_features_to_select=5)
rfe.fit(X_train, y_train)
ranking_ 속성에는 선택한 특성의 우선순위가 들어 있다.
(1은 기반 모델이 선택한 5개의 특성)
array([1, 5, 9, 6, 4, 3, 1, 8, 7, 1, 2, 1, 1])
선택한 특성의 정보는 support_에 들어 있다.
선택한 기반모델(랜덤 포레스트 모델)은 estimator_속성에 저장되어 있다.
이 모델은 n_feature_to_select개의 특성을 이용하여, 훈련되어 있다.
importances=rfe.estimator_.feature_importances_
indices=np.argsort(importances)[::-1]
for i in indices:
print("%2d) %-*s %f" %(f+1, 30, feat_labels[f_mask][i], importances[i]))
5) Proline 0.261512
5) Color intensity 0.216477
5) Flavanoids 0.212259
5) 0D280/0D315 of diluted wines 0.188924
5) Alchol 0.120828